本項でもGoogle Colaboratoryを使用する.(もちろんファイル読み込みの部分以外はJupyter Notebookでもよいし,一気にPythonのソースコードをまとめて書いて実行しても問題はない)
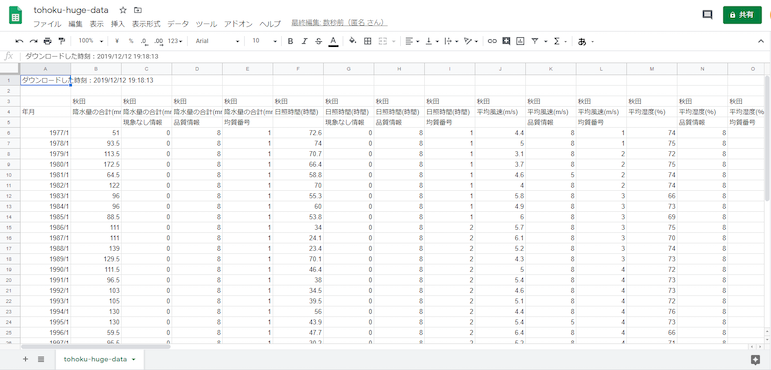
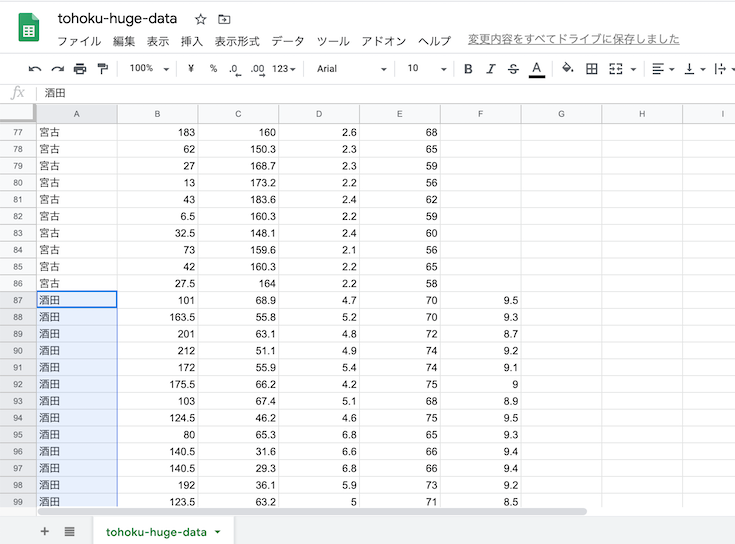
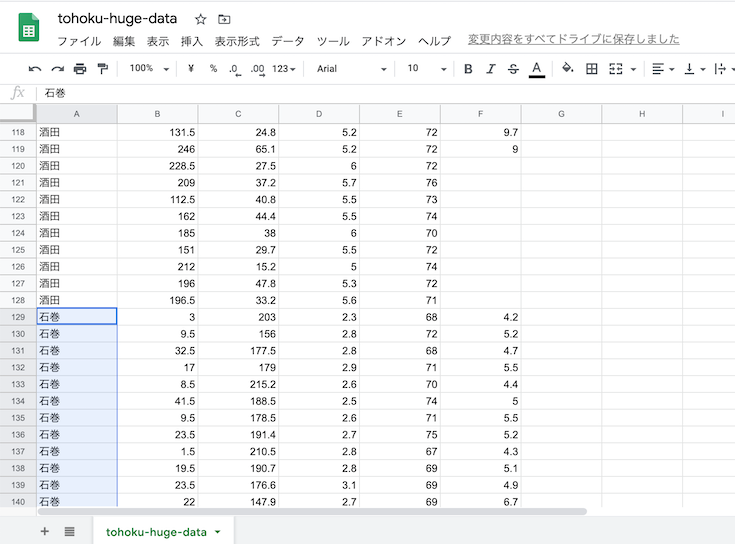
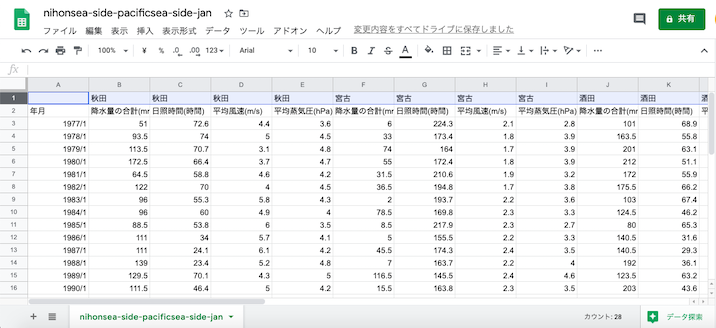
今回も「気温以外のデータ」を使って,さらに線形分類の時よりも観測データを多くして,太平洋側の都市と日本海側の都市を比較してみる.
例によって,気象庁オープンデータのページから,
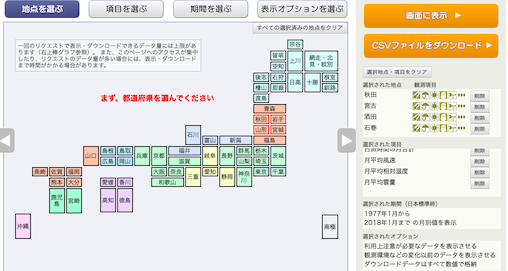
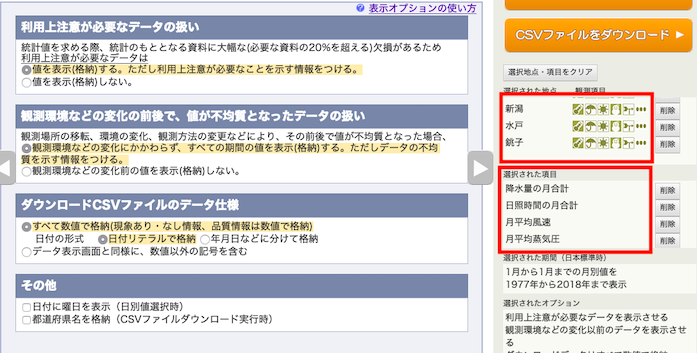
都市は,秋田県→秋田市,岩手県→宮古市,山形県→酒田市,宮城県→石巻市,の順で,それぞれ選択.
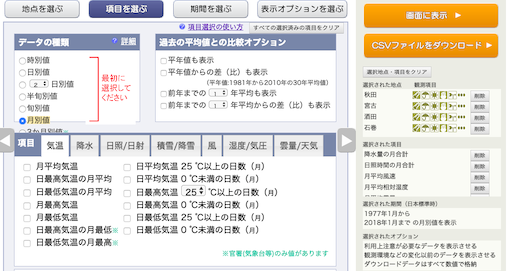
今回も「気温以外のデータ」なので,気温は選択しない.
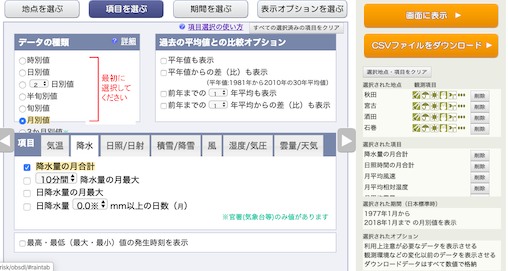
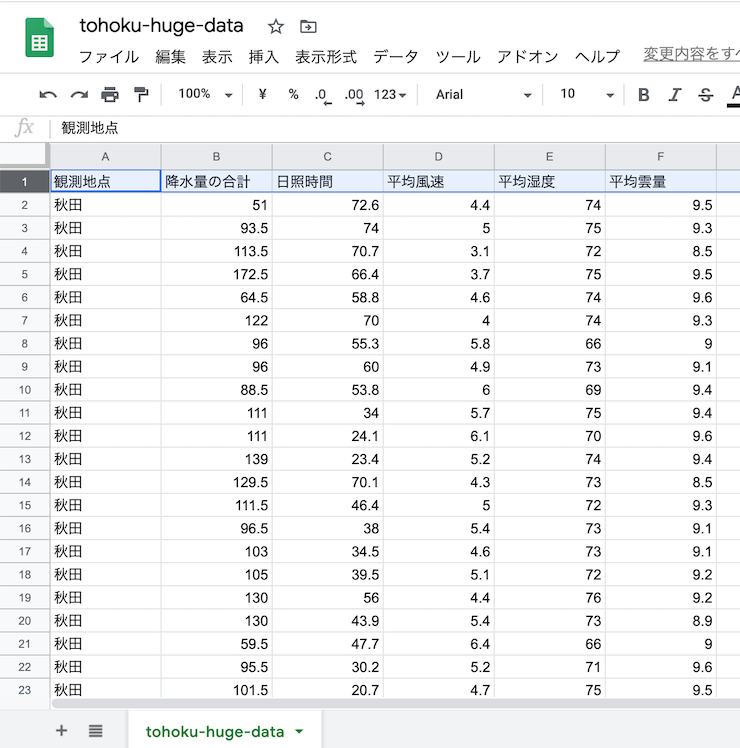
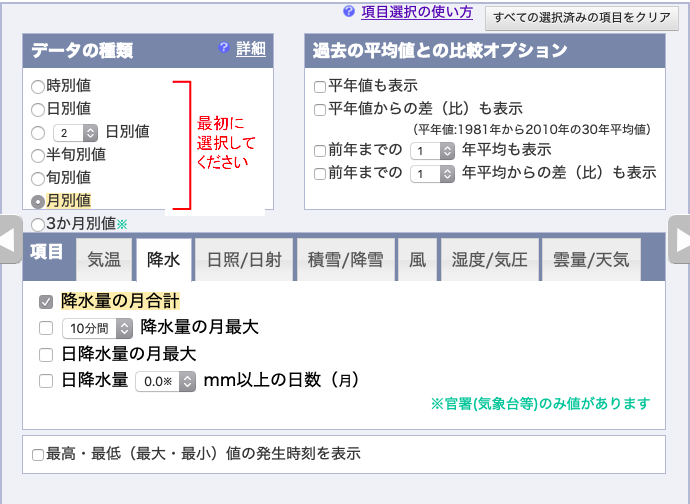
月別の値を選択し,降水量の月合計,
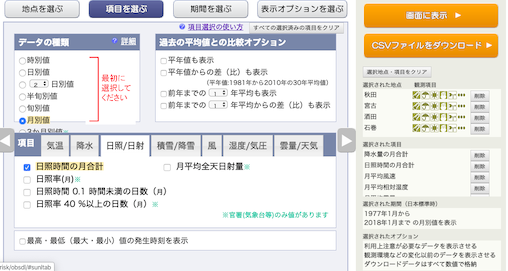
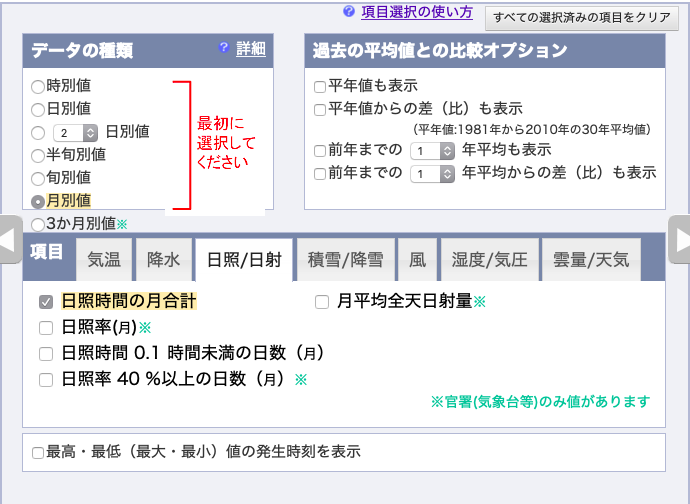
日照時間の月合計,
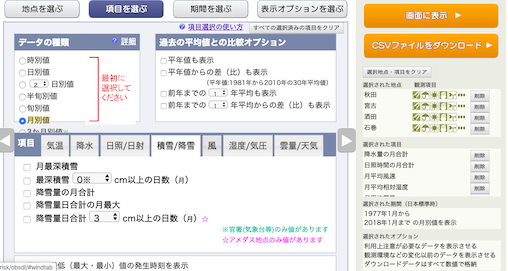
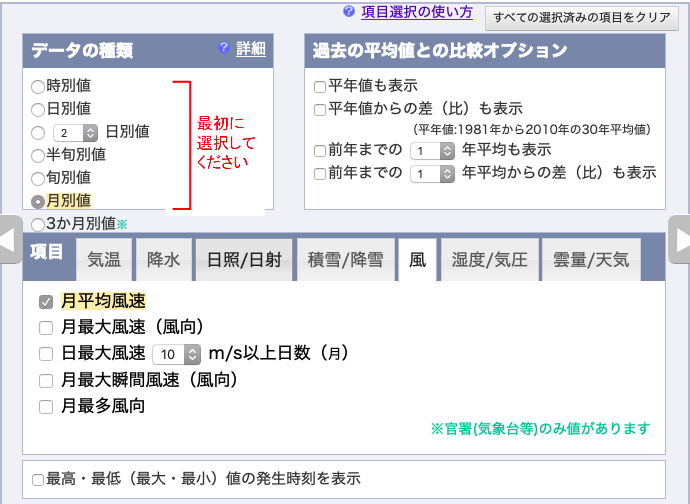
月平均風速,
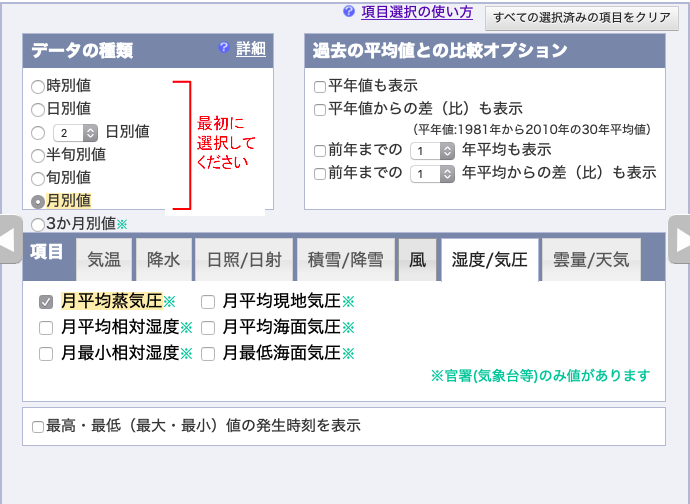
湿度/気圧の月平均蒸気圧,を選択し,
平均雲量,を選択し,
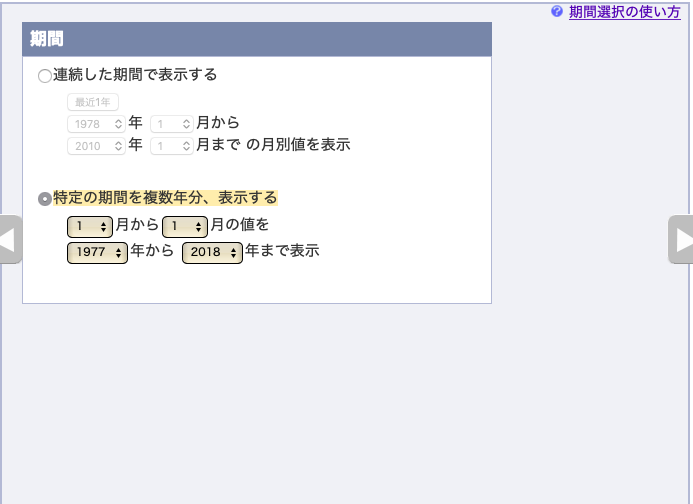
1977年〜2018年の「1月」のデータをダウンロードする.
表示オプションはデフォルト.地点の順番と,項目の順番を確認すること.
因子分析
因子分析とは,観測された変数の「原因となっている何らかの変数」が隠れて存在する(潜在変数)と考え,その変数を探す分析のことである.
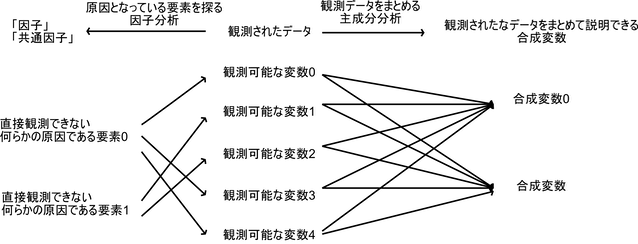
例えば太平洋側の都市と日本海側の都市では冬の気候に大きな違いがあるが,観測データには何らかの「観測可能なデータの違い」があり,その観測可能なデータから「原因」である「気候の違い」を,「なるべく少ない要素」で表現できるように探すことが目的である.
「どちらか側の海」というのは根本的な原因であるダミー変数だが,ダミー変数を直接出せるほど因子分析は優れてはいないので,潜在変数はスカラ値であることがほとんどである.
次元圧縮としても使えてしまうため,主成分分析と混同されることがあるが,因子分析と主成分分析は考え方が逆である.
因子分析は「観測可能なデータから,観測データの原因となっている少数の要素(因子,潜在変数)があると仮定して,それを推測する」
主成分分析は「観測可能なデータから,少数で多くの観測データを上手く表現することができる少数の合成変数を作成する」
である.
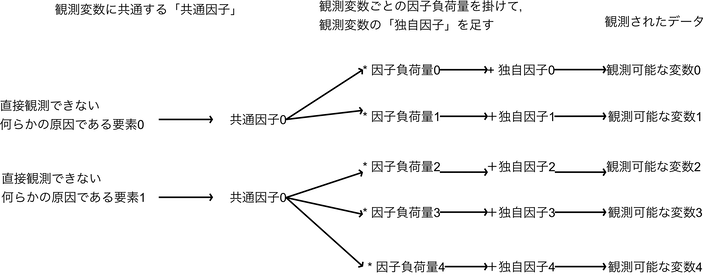
実際には「共通因子」に,どのぐらい共通因子が占める割合が大きいかの「因子負荷量」を掛け,その変数の独自の要素である「独自因子」を足したものが,観測データである.
因子分析の特徴として,因子の数がいくつあるかを与えなければならない.因子分析の手法は多く提唱されており色々な組み合わせがあるが,「回転法」が直交法 - Varimax回転の時に因子数の指標となるのが「因子負荷量」である.
因子負荷量は,その仮定した因子がどのぐらい各観測変数に影響を与えているか(正確には各観測変数と各因子の相関係数,つまり連動して動いているか)を見るもので,因子負荷量が各変数において各因子間の違いが大きければ大きいほど,その仮定した因子数は適切である,と考える.(相関係数なので-1から1の間に収まる.0の時は「観測変数とその因子は全く連動していない」ということになる.)
具体的には,「各変数の各因子に対する因子負荷量を見て,一つの因子のみ1(もしくは-1)に近く,他の因子は0に近い」場合には,その因子の選定は適切である,となる.
コード例
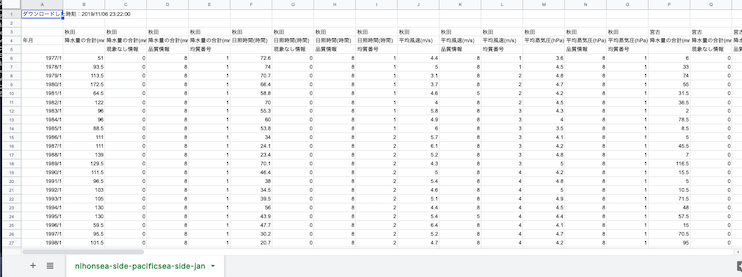
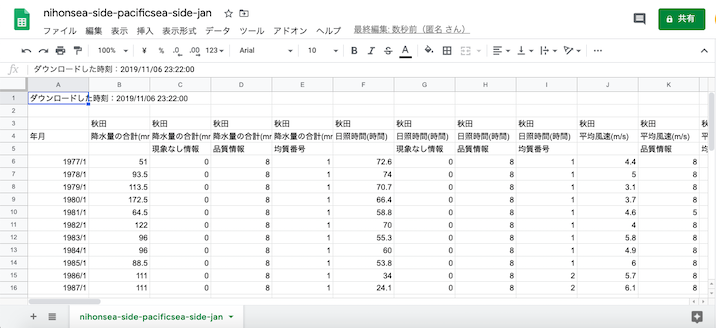
1つ目のセル.いつもと同じくCSVをColabへ読み込む.
from google.colab import files
uploaded = files.upload()
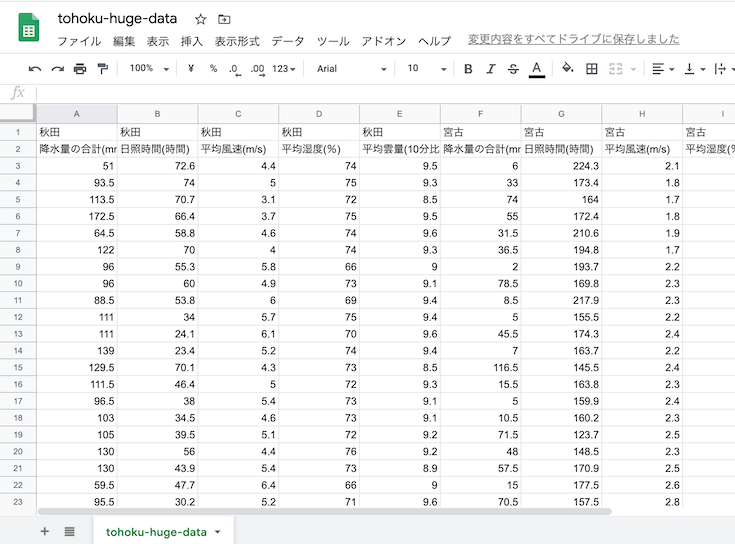
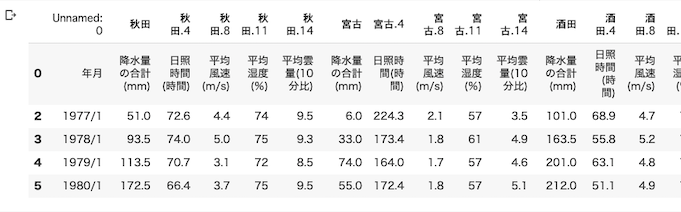
2つ目のセル.品質情報などの列が不要なので削除.
# CSVを整えてDataFrameにする
import numpy as np
import pandas as pd
from pandas import Series, DataFrame
df = pd.read_csv('tohoku-huge-data.csv', encoding='SHIFT-JIS', header=1)
# 抜き出す
for a_column in ['秋田', '宮古', '酒田', '石巻']:
# それぞれ
# 降水量の合計(mm),降水量の合計(mm),降水量の合計(mm),降水量の合計(mm),日照時間(時間),日照時間(時間),日照時間(時間),日照時間(時間),平均風速(m/s),平均風速(m/s),平均風速(m/s),平均湿度(%),平均湿度(%),平均湿度(%),平均雲量(10分比),平均雲量(10分比),平均雲量(10分比)
# で並んでいて,一つの地点につき,
# (空),現象なし情報,品質情報,均質番号,(空),現象なし情報,品質情報,均質番号,(空),品質情報,均質番号,(空),品質情報,均質番号,(空),品質情報,均質番号
# の順に並んでいる
# の(空)以外の場所が余計なので,削除
for i in range(1, 17, 1):
# 落とす列の列番号を指定(0~)
if i in [1, 2, 3, 5, 6, 7, 9, 10, 12, 13, 15, 16]:
df = df.drop(a_column + '.' + str(i), axis=1)
df = df.drop(1)
# DataFrameの中に欠損値がある場合は,0.0を代わりに入れておく
df = df.fillna(0)
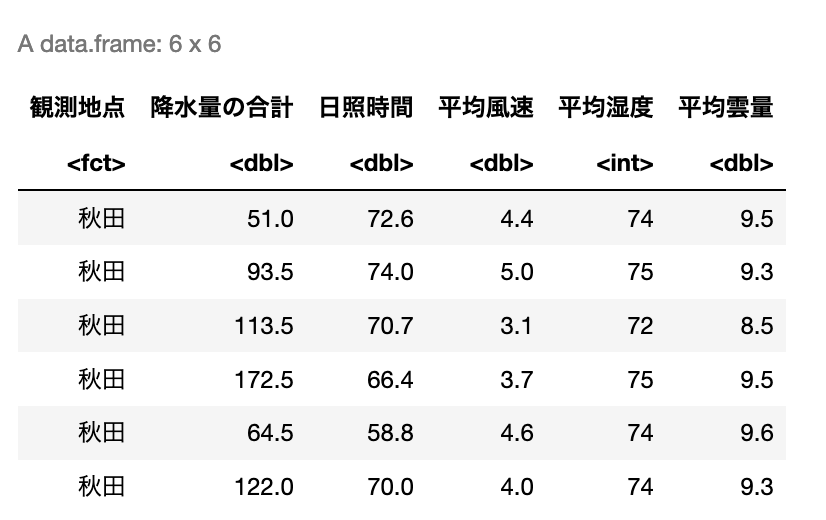
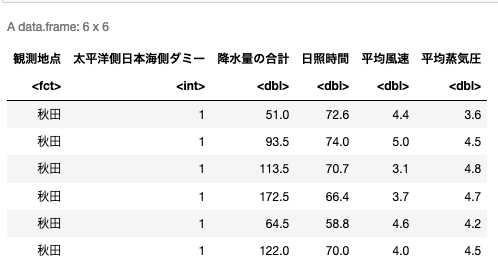
df.head()2つ目のセルの結果は以下の通り
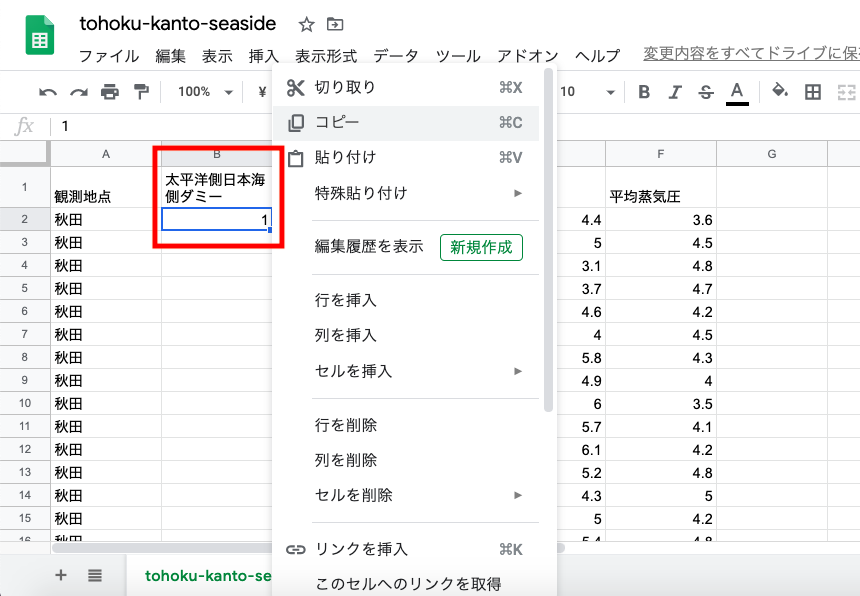
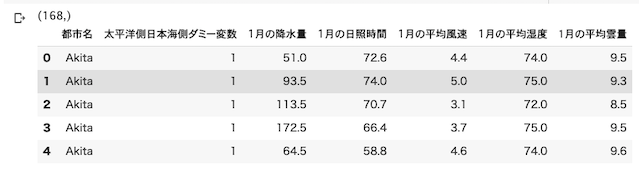
3つ目のセル.都市名,ダミー変数を入れて,各都市のデータの列を繋いで,一つのデータフレームにする.
# グラフ表示するためにローマ字ラベルを作っておく
#np.arrayはenumerateでは回せないので, listのままにしておく
city_label_list = (['Akita', 'Miyako', 'Sakata', 'Ishinomaki'])
# その要素の有無で0と1の2値で表現される変数を「ダミー変数」と呼ぶ
# 0, 1, 2, 3...などの状態が複数あり,かつその数字が大小の意味を持ってない場合は,カテゴリ変数と呼ぶ
# カテゴリ変数は判別問題などで扱えないので,
# カテゴリ変数で表現している内容を,要素に分けて,要素の有無で表現,つまり複数のダミー変数に直す
# 今回は太平洋側と日本海側の2値なので,ダミー変数:太平洋側が0, 日本海側が1
sea_side_label = np.array([1, 0, 1, 0])
# 選んだ都市の
# precipitation: 1月の降水量
# daylight_hours: 1月の日照時間
# average_wind_speed: 1月の平均風速
# average_humidity: 1月の平均湿度
# amounts_clouds: 1月の平均雲量
# として,それぞれ42年分*太平洋側,日本海側の2年ずつを,1つ配列としてくっつける
# 42年分のラベルを繋げておく (42年分*全部の都市)
city_label_in_repeat = np.repeat(city_label_list, 42)
sea_side_label_in_repeat = np.repeat(sea_side_label, 42)
print(sea_side_label_in_repeat.shape)
# データをつなげる
# まず最初に空の配列を作っておいて,
precipitation = np.array([], dtype=np.float)
daylight_hours = np.array([], dtype=np.float)
average_wind_speed = np.array([], dtype=np.float)
average_humidity = np.array([], dtype=np.float)
amounts_clouds = np.array([], dtype=np.float)
# 選んだ都市の,1月の平均風速,1月の日照時間,1月の平均湿度,1月の降水量を
# それぞれ1つの配列につなぐ
for i, a_city in enumerate(city_label_list):
precipitation = np.concatenate((precipitation, df.iloc[1:, 1+(i*5)]))
daylight_hours = np.concatenate((daylight_hours, df.iloc[1:, 1+(i*5)+1]))
average_wind_speed = np.concatenate((average_wind_speed, df.iloc[1:, 1+(i*5)+2]))
average_humidity = np.concatenate((average_humidity, df.iloc[1:, 1+(i*5)+3]))
amounts_clouds = np.concatenate((amounts_clouds, df.iloc[1:, 1+(i*5)+4]))
# vstackで重ねて,配列の次元を入れ替えて,DataFrameにしておく
df_sea_side_city = pd.DataFrame(np.vstack([city_label_in_repeat, sea_side_label_in_repeat, precipitation.astype(np.float), daylight_hours.astype(np.float), average_wind_speed.astype(np.float), average_humidity.astype(np.float), amounts_clouds.astype(np.float)]).transpose())
# DataFrameの列名をつけておく
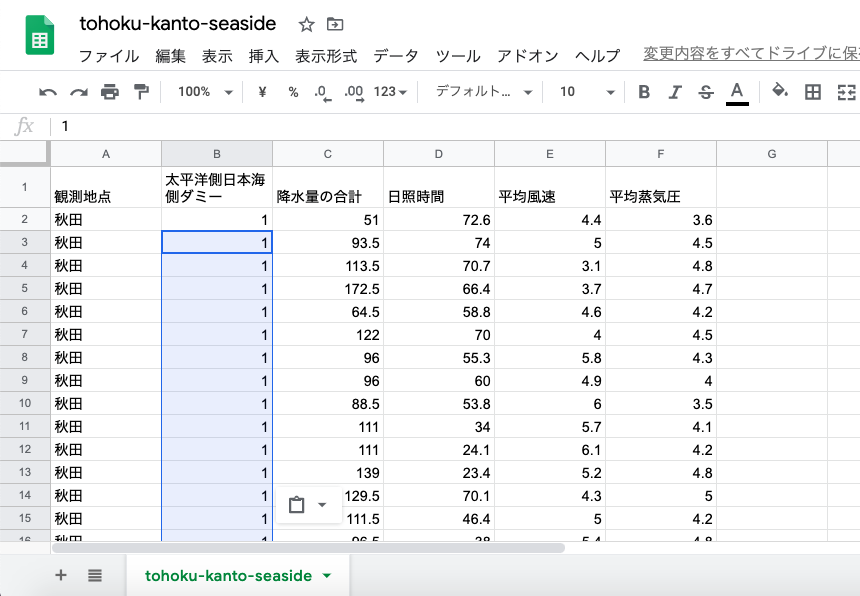
df_sea_side_city = df_sea_side_city.rename(columns={0:'都市名', 1:'太平洋側日本海側ダミー変数', 2:'1月の降水量', 3:'1月の日照時間', 4:'1月の平均風速', 5:'1月の平均湿度', 6:'1月の平均雲量'})
# DataFrameの冒頭を確認
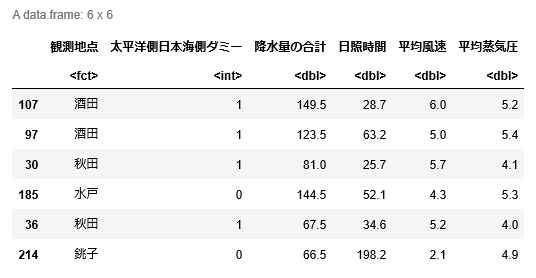
df_sea_side_city.head()3つ目のセルの結果は以下の通り
4つ目のセル.因子分析のパッケージをインストールする.
# 高機能因子分析パッケージ
!pip install factor-analyzer5つ目のセル.変数の数が5つなので,とりあえず因子数4で因数分解をして,因子負荷量を算出してみる.
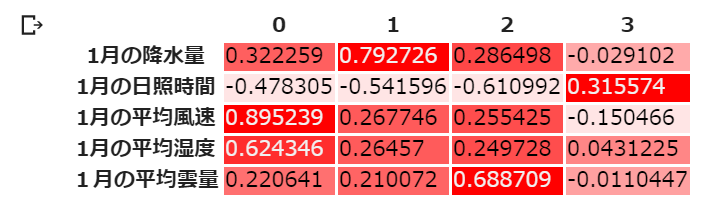
各共通因子と各観測変数の間の「因子負荷量」である.因子負荷量は「その因子が変数にどれだけ影響を与えているか」の値で,-1から1の間を取る.
1つの観測変数に対する因子負荷量が各因子間(ここではML1とML2~4の間)で大きく差がある(典型的なのは,1観測変数に対して,1因子だけ1もしくは-1に近く,他の因子では0に近い)と,うまく因子分析が機能し共通因子にまとめられたことを表し,逆に.1つの観測変数に対して因子負荷量の因子間の差が小さい場合は因子分析がうまくいかなかったことを示している.
# 因子分析
# 観測されたデータ(説明変数)が大量の次元を持ち,
# かつ説明変数感の相関係数が高い場合,
# 因果のルート側に数が少ない潜在変数Latent Variable(因子 Factor)が存在することにより,
# 観測データ = 説明変数が生み出されているという考え方
# 回帰や分類の前処理に使う場合の目的は,次元圧縮なので主成分分析と同じに思えるが,
# 因子分析は,観測データ(説明変数)を潜在変数の組み合わせによる合成量として捉え,その潜在変数を求める
# つまり考え方は主成分分析の逆である.
# 次元圧縮を目的としている場合,次元数を与えられるのは有利だが,
# 機械学習目的ではなく,分析目的の場合,
# 潜在変数の数,すなわち次元数を最初に与えなければならないことは,欠点でもある
# 次元圧縮した状態でグラフ等をみて,これは何の因子なのか,因子が妥当かどうか,を
# 分析者が検討しなければならい
from factor_analyzer import FactorAnalyzer
import seaborn as sns
# 何因子だと考えるか
# とりあえず最初は多めに
n_factors=4
fa_model = FactorAnalyzer(n_factors=n_factors, method='ml', rotation='varimax', impute='drop')
# method: 使用する手法
# minres (ミンレス法)
# ml (最尤法)
# principal(主因子法)
# rotation 回転法
# varimax (バリマックス回転) 直交法
# promax (プロマックス回転) 斜交法
# oblique (斜め回転)
# impute 欠損値をどうするか
# drop: 削除
# データにフィットさせる
# 因子分析の対象にするのは5観測変数であり,都市名と太平洋側に日本海側ダミー変数は使わない
fa_model.fit(df_sea_side_city.iloc[:, 2:])
# 因子負荷量はloadings_プロパティで読み出せるので,結果のDataFrameを作る
# 因子負荷量:因子と観測変数の間の関係性の強弱のこと
# わかりやすく列名を,元のデータフレームから持ってきて挿入する
# 0列目はラベルなので,1列目から
columns_name_array = df_sea_side_city.columns[2:]
# 行名(index)として,この配列を用いて,DataFrameを作る
df_result = pd.DataFrame(fa_model.loadings_, index=columns_name_array)
# 因子負荷量の行列を色分けして表示
cm = sns.light_palette('red', as_cmap=True
# セルの最後に実行すること.df.head()と同じく,printしても色分けされない
df_result.style.background_gradient(cmap=cm)5つ目のセルの結果は以下の通り.
Varimax回転をPromax回転に変えるなどして,回転方法によって結果が変わることを確かめる.(ただしPromax回転の場合因子負荷量に意味はないので参考程度に)
因子分析が「うまくいった」と判断される因子負荷量は「1観測変数に対して,1共通因子だけ1もしくは-1に近く,他の共通因子では0に近い」である.逆に.1つの観測変数に対して因子負荷量の共通因子間の差が小さい場合は因子分析がうまくいかなかったことを示している.
この共通因子数4を仮定した例では,日照時間に関して,負の方向では因子間の違いがあまりない(共通因子2と4の間がほとんど差がない)ことから,因子分析がうまくいっておらず,共通因子数4を仮定するのは,あまり具合が良くない,と言うことがわかる.
6つ目のセル.Varimax回転を使っている場合,累積寄与率を出すことができる.寄与率は主成分分析の時と同じく,「設定した因子で観測データをどのぐらい説明できるか」の基準である.6つ目のセルではこの累積を算出する.
# Varimax回転の場合の因子数4での累積寄与率
factor_index = list([])
for i in range(n_factors):
factor_index.append('因子' + str(i))
df_cumulative_variance = pd.DataFrame(fa_model.get_factor_variance()[2], index=factor_index)
df_cumulative_variance.style.background_gradient(cmap=cm)6つ目のセルの出力結果は以下の通り
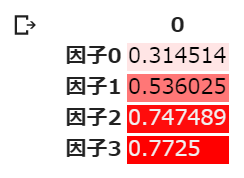
Varimax回転の場合は,累積寄与率を参照することができる.(Promax回転場合は,累積寄与率は出ても値に意味はない.寄与率そのものは相対的に扱うことができる)
3因子で全体の74.7%,4因子で全体の77.2%を「説明することができている」
因子3の寄与率が高くないので,やはり因子数が4の設定では多すぎる,と判断することができる.
7つ目のセルでは因子数4の時の,観測値を共通因子に変換し,その散布図を描く.共通因子数4次元なのでそれぞれの因子を軸にとると,3*2で6つのグラフを描くことができる.
# 因子分析の結果を使って,
# 観測された元々の説明変数の値を潜在変数の値に変換する
factors_df = pd.DataFrame(fa_model.transform(df_sea_side_city.iloc[:, 2:]))
num_of_row = df_sea_side_city.values.shape[0]
# 潜在変数の値をプロットしてみる
akita = list([])
miyako = list([])
sakata = list([])
kamaishi = list([])
for i in range(num_of_row):
if df_sea_side_city.values[i][0] == 'Akita':
akita.append(factors_df.iloc[i, :])
elif df_sea_side_city.values[i][0] == 'Miyako':
miyako.append(factors_df.iloc[i, :])
elif df_sea_side_city.values[i][0] == 'Sakata':
sakata.append(factors_df.iloc[i, :])
else:
kamaishi.append(factors_df.iloc[i, :])
df_akita = pd.DataFrame(akita)
df_miyako = pd.DataFrame(miyako)
df_sakata = pd.DataFrame(sakata)
df_kamaishi = pd.DataFrame(kamaishi)
import matplotlib.pyplot as plt
plt.scatter(df_akita.iloc[:, 0], df_akita.iloc[:, 1], label='Akita')
plt.scatter(df_sakata.iloc[:, 0], df_sakata.iloc[:, 1], label='Sakata')
plt.scatter(df_miyako.iloc[:, 0], df_miyako.iloc[:, 1], label='Miyako')
plt.scatter(df_kamaishi.iloc[:, 0], df_kamaishi.iloc[:, 1], label='Kamaishi')
plt.xlabel('Factor0')
plt.ylabel('Factor1')
plt.legend()
plt.show()
plt.scatter(df_akita.iloc[:, 0], df_akita.iloc[:, 2], label='Akita')
plt.scatter(df_sakata.iloc[:, 0], df_sakata.iloc[:, 2], label='Sakata')
plt.scatter(df_miyako.iloc[:, 0], df_miyako.iloc[:, 2], label='Miyako')
plt.scatter(df_kamaishi.iloc[:, 0], df_kamaishi.iloc[:, 2], label='Kamaishi')
plt.xlabel('Factor0')
plt.ylabel('Factor2')
plt.legend()
plt.show()
plt.scatter(df_akita.iloc[:, 0], df_akita.iloc[:, 3], label='Akita')
plt.scatter(df_sakata.iloc[:, 0], df_sakata.iloc[:, 3], label='Sakata')
plt.scatter(df_miyako.iloc[:, 0], df_miyako.iloc[:, 3], label='Miyako')
plt.scatter(df_kamaishi.iloc[:, 0], df_kamaishi.iloc[:, 3], label='Kamaishi')
plt.xlabel('Factor0')
plt.ylabel('Factor1')
plt.legend()
plt.show()
plt.scatter(df_akita.iloc[:, 1], df_akita.iloc[:, 2], label='Akita')
plt.scatter(df_sakata.iloc[:, 1], df_sakata.iloc[:, 2], label='Sakata')
plt.scatter(df_miyako.iloc[:, 1], df_miyako.iloc[:, 2], label='Miyako')
plt.scatter(df_kamaishi.iloc[:, 1], df_kamaishi.iloc[:, 2], label='Kamaishi')
plt.xlabel('Factor1')
plt.ylabel('Factor2')
plt.legend()
plt.show()
plt.scatter(df_akita.iloc[:, 1], df_akita.iloc[:, 3], label='Akita')
plt.scatter(df_sakata.iloc[:, 1], df_sakata.iloc[:, 3], label='Sakata')
plt.scatter(df_miyako.iloc[:, 1], df_miyako.iloc[:, 3], label='Miyako')
plt.scatter(df_kamaishi.iloc[:, 1], df_kamaishi.iloc[:, 3], label='Kamaishi')
plt.xlabel('Factor1')
plt.ylabel('Factor3')
plt.legend()
plt.show()
plt.scatter(df_akita.iloc[:, 2], df_akita.iloc[:, 3], label='Akita')
plt.scatter(df_sakata.iloc[:, 2], df_sakata.iloc[:, 3], label='Sakata')
plt.scatter(df_miyako.iloc[:, 2], df_miyako.iloc[:, 3], label='Miyako')
plt.scatter(df_kamaishi.iloc[:, 2], df_kamaishi.iloc[:, 3], label='Kamaishi')
plt.xlabel('Factor2')
plt.ylabel('Factor3')
plt.legend()
plt.show()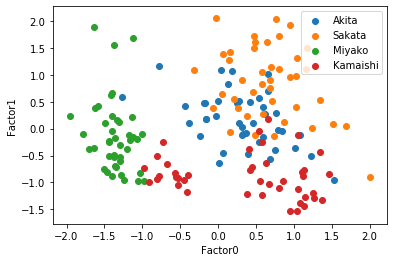
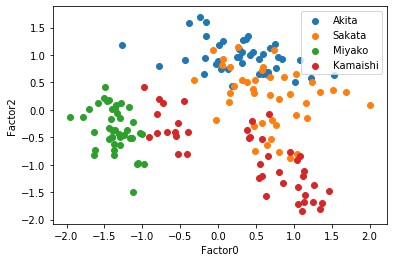
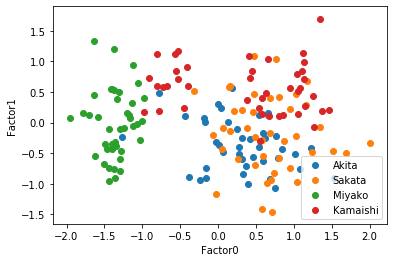
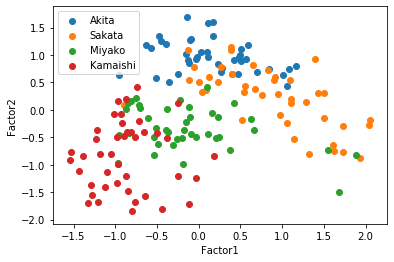
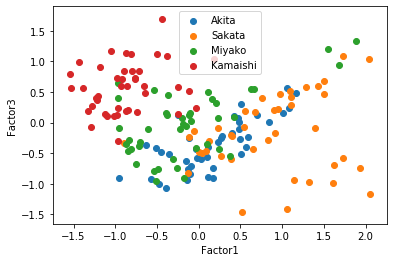
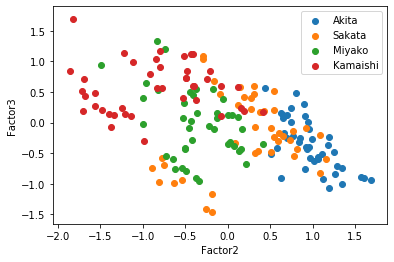
この散布図を見ると,因子1-2の散布図,因子1-3の両方で太平洋側と日本海側を直線で分けられるように見える,因子負荷量を考えると,もっと因子数が少ない方がいいかもしれない.そこで因子数3と2についてもまったく同じように行っていく.
8つ目のセル.因子数3に設定して,まったく同じように因子分析を行い,因子負荷量を出す.
# 因子数3
n_factors=3
fa_model = FactorAnalyzer(n_factors=n_factors, method='ml', rotation='varimax', impute='drop')
fa_model.fit(df_sea_side_city.iloc[:, 2:])
# わかりやすく列名を,元のデータフレームから持ってきて挿入する
# 0列目はラベルなので,1列目から
columns_name_array = df_sea_side_city.columns[2:]
# 行名(index)として,この配列を用いて,DataFrameを作る
df_result = pd.DataFrame(fa_model.loadings_, index=columns_name_array)
# 因子負荷量の行列を色分けして表示
cm = sns.light_palette('red', as_cmap=True)
# セルの最後に実行すること.df.head()と同じく,printしても色分けされない
df_result.style.background_gradient(cmap=cm)8つ目のセルの結果は以下の通り.
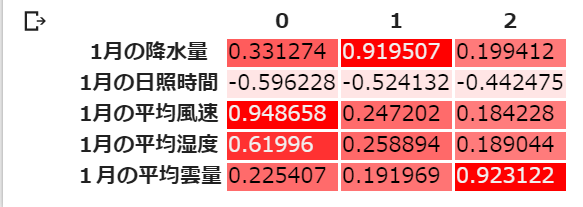
日照時間の因子負荷量の因子間の違いが,因子0と因子1間で0.07, 因子1と因子2間で0.08と,非常に小さい.これは因子数3では因子分析がうまくいかなかったことを示している.
9つ目のセルでは,一応累積寄与率も出してみる.(前述のようにVarimax回転の時に有効.Promax回転の時は無効)
# Varimax回転の時の因子数3での累積寄与率
# Varimax回転の場合は,累積寄与率を参照することができる.
factor_index = list([])
for i in range(n_factors):
factor_index.append('因子' + str(i))
df_cumulative_variance = pd.DataFrame(fa_model.get_factor_variance()[2], index=factor_index)
df_cumulative_variance.style.background_gradient(cmap=cm)9つ目のセルの実行結果は以下の通り.
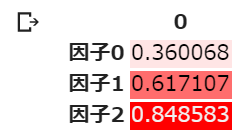
3因子の累積寄与率が84.9%なので,因子数3で因子分析を行った方が全体をうまく説明できていることになるが,因子負荷量の差が小さいことを考えると,総合的にはよくない.
10個目のセルは因子数2に設定して,まったく同じように,因子分析を行い,因子負荷量を出している.
# 因子数2
n_factors=2
fa_model = FactorAnalyzer(n_factors=n_factors, method='ml', rotation='varimax', impute='drop')
fa_model.fit(df_sea_side_city.iloc[:, 2:])
# わかりやすく列名を,元のデータフレームから持ってきて挿入する
# 0列目はラベルなので,1列目から
columns_name_array = df_sea_side_city.columns[2:]
# 行名(index)として,この配列を用いて,DataFrameを作る
df_result = pd.DataFrame(fa_model.loadings_, index=columns_name_array)
# 因子負荷量の行列を色分けして表示
cm = sns.light_palette('red', as_cmap=True)
# セルの最後に実行すること.df.head()と同じく,printしても色分けされない
df_result.style.background_gradient(cmap=cm)10個目のセルの実行結果は以下の通り.
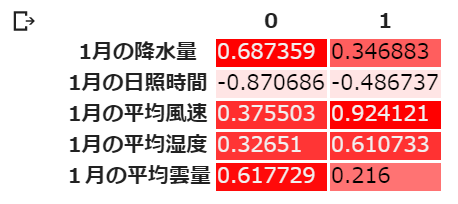
因子数2の時,因子負荷量が因子間でそれなりに大きく離れている.降水量,日照時間,雲量は因子0に,風速と湿度は因子1にそれぞれ大きな影響を受けている.
因子数を3以下にする場合因子数2が適切っぽい.確かめるために散布図を描いてみる.
11個目のセルでは因子数2の時の散布図を描く.因子数2なので2次元グラフ1つ.
# 因子分析の結果を使って,
# 観測された元々の説明変数の値を潜在変数の値に変換する
factors_df = pd.DataFrame(fa_model.transform(df_sea_side_city.iloc[:, 2:]))
num_of_row = df_sea_side_city.values.shape[0]
# 潜在変数の値をプロットしてみる
akita = list([])
miyako = list([])
sakata = list([])
kamaishi = list([])
for i in range(num_of_row):
if df_sea_side_city.values[i][0] == 'Akita':
akita.append(factors_df.iloc[i, :])
elif df_sea_side_city.values[i][0] == 'Miyako':
miyako.append(factors_df.iloc[i, :])
elif df_sea_side_city.values[i][0] == 'Sakata':
sakata.append(factors_df.iloc[i, :])
else:
kamaishi.append(factors_df.iloc[i, :])
df_akita = pd.DataFrame(akita)
df_miyako = pd.DataFrame(miyako)
df_sakata = pd.DataFrame(sakata)
df_kamaishi = pd.DataFrame(kamaishi)
import matplotlib.pyplot as plt
plt.scatter(df_akita.iloc[:, 0], df_akita.iloc[:, 1], label='Akita')
plt.scatter(df_sakata.iloc[:, 0], df_sakata.iloc[:, 1], label='Sakata')
plt.scatter(df_miyako.iloc[:, 0], df_miyako.iloc[:, 1], label='Miyako')
plt.scatter(df_kamaishi.iloc[:, 0], df_kamaishi.iloc[:, 1], label='Kamaishi')
plt.xlabel('Factor0')
plt.ylabel('Factor1')
plt.legend()
plt.show()11個目のセルの実行結果は以下の通り.
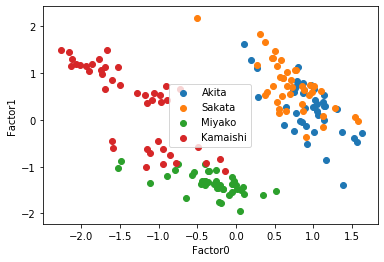
因子数2だと日本海側と太平洋側で直線分離できるので,とりあえずこれで因子に名前を付けることにする.
因子1は平均風速と湿度に正の大きな影響与えている.かつ風速の方が影響が大きい.これは「荒天度」ということができるだろう.
では「降水量」「日照時間」「雲量」に影響を大きく与えている因子0にはどのような名前をつければよいだろうか.日照時間は負の影響であることに注目して名前を付けてみよう.
12個目のセルは以下の通りである.因子数2の時の「共通性」,つまり各観測変数がどれだけ共通因子に影響を受けているかを出す.
# 共通性
# 共通性とは,観測可能な変数における共通因子が占める割合のこと.
# これが低くなると独自因子の割合が多くなる.
# つまり共通因子とどれだけ連動しているかの値を見ることができる
df_communalities = pd.DataFrame(fa_model.get_communalities(), index=columns_name_array)
df_communalities.style.background_gradient(cmap=cm)12個目のセルの実行結果は以下の通り.
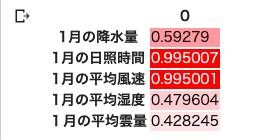
日照時間と風速の値が大きいので,日照時間は因子0の影響を降水量や雲量よりも大きく受けている,風速は因子1の影響を湿度よりも大きく受けている,と言うことができる.
逆に,降水量や雲量,湿度は,日照時間や風速に比べ「共通因子よりも,固有の独自因子の影響を多く受けている」と言うこともできる.
この共通性が非常に小さい時,その観測変数は共通因子から受ける影響が非常に少ないということであり,この観測変数を外して再度因子分析をかけるのが適切である.
「1 - 共通性」の値が「独自性」であり,その変数が共通因子から受けている影響の小ささを表す.
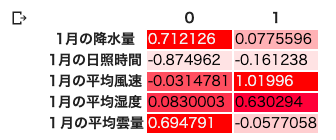
13個目のセルでは,因子数2として,Promax回転の因子分析を行う.
Promax回転(斜交回転)は「因子間に相関性がある」ことを前提とした手法であり,「因子間に相関性がないなんてことは,状況的に考えてそうはない」場合にはこちらを用いる.
# 因子数2のプロマックス回転
n_factors=2
fa_model = FactorAnalyzer(n_factors=n_factors, method='ml', rotation='promax', impute='drop')
fa_model.fit(df_sea_side_city.iloc[:, 2:])
columns_name_array = df_sea_side_city.columns[2:]
df_result = pd.DataFrame(fa_model.loadings_, index=columns_name_array)
cm = sns.light_palette('red', as_cmap=True)
df_result.style.background_gradient(cmap=cm)13個目のセルの実行結果は以下の通り.プロマックス回転では因子負荷量行列ではなく「パターン行列」と呼ぶ.
14個目のセルでは,バリマックス回転と全く同じように,潜在変数の散布図を描いた.
# 因子分析の結果を使って,
# 観測された元々の説明変数の値を潜在変数の値に変換する
factors_df = pd.DataFrame(fa_model.transform(df_sea_side_city.iloc[:, 2:]))
num_of_row = df_sea_side_city.values.shape[0]
# 潜在変数の値をプロットしてみる
akita = list([])
miyako = list([])
sakata = list([])
kamaishi = list([])
for i in range(num_of_row):
if df_sea_side_city.values[i][0] == 'Akita':
akita.append(factors_df.iloc[i, :])
elif df_sea_side_city.values[i][0] == 'Miyako':
miyako.append(factors_df.iloc[i, :])
elif df_sea_side_city.values[i][0] == 'Sakata':
sakata.append(factors_df.iloc[i, :])
else:
kamaishi.append(factors_df.iloc[i, :])
df_akita = pd.DataFrame(akita)
df_miyako = pd.DataFrame(miyako)
df_sakata = pd.DataFrame(sakata)
df_kamaishi = pd.DataFrame(kamaishi)
import matplotlib.pyplot as plt
plt.scatter(df_akita.iloc[:, 0], df_akita.iloc[:, 1], label='Akita')
plt.scatter(df_sakata.iloc[:, 0], df_sakata.iloc[:, 1], label='Sakata')
plt.scatter(df_miyako.iloc[:, 0], df_miyako.iloc[:, 1], label='Miyako')
plt.scatter(df_kamaishi.iloc[:, 0], df_kamaishi.iloc[:, 1], label='Kamaishi')
plt.xlabel('Factor0')
plt.ylabel('Factor1')
plt.legend()
plt.show()14個目のセルの実行結果は以下の通り.斜交回転だと軸に沿って出力されるのがわかる.
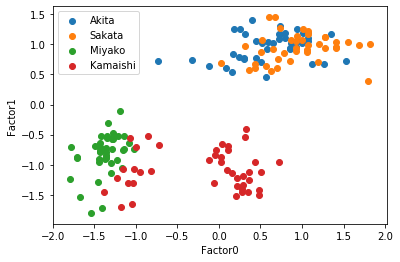
プロマックス回転だと,日本海側と太平洋側がはっきり分離する.したがってこの場合「因子1は太平洋側と日本海側」だと考えることができそうだ.
また,日本海側の都市はPromax回転でも分離して見えないが,宮古と釜石は分離して見えるようになる.因子0が宮古と釜石の違いであると言えそうだ.さて因子0はなんと名前をつければいいだろうか.考えてみよう.